Компания «Лаборатория эпигенетической ДНК диагностики» занимается разработкой нового поколения эпигенетических ПЦР тестов для выявления онкологических заболеваний на ранних стадиях.
В наших тестах осуществляется анализ ДНК из венозной крови с использованием стандартного оборудования для ПЦР в реальном времени.
Что делает наш подход особенным?
Мы анализируем маркеры метилирования ДНК, считающиеся наиболее перспективными для выявления рака на ранних стадиях, но используем собственный абсолютно новый метод анализа метилирования ДНК.
Анализ основан на использовании нового типа ферментов, ранее открытого командой ученых компании SibEnzyme — метил-зависимых ДНК эндонуклеазах или MD ДНК эндонуклеазах. Новые ферменты по своим свойствам очень похожи на обычные эндонуклеазы рестрикции, но в противоположность им режут только метилированную ДНК. Это делает их удобным инструментом для эпигенетических исследований. Дополнительная информация
Два абсолютно новых метода анализа ДНК: GLAD-ПЦР анализ и GlaI-ПЦР-анализ — для эпигетического анализа препаратов ДНК из клинических образцов. Позволяет определять наличие даже нескольких молекул метилированной ДНК (сайта R(5mC)GY) или неметилированной ДНК (сайта RCGY) соответственно в образце с избыточном количеством неметилированной или метилированной ДНК.
Дополнительная информация: GLAD-ПЦР, GlaI-ПЦР
Фрагментация геномной ДНК ферментом GlaI с последующим NGS позволяет строить профили метилирования для больных и здоровых доноров. Сравнение данных профилей позволяет с легкостью находить сайты R(5mC)GY — потенциальные эпигенетические маркеры, которые могут использоваться в диагностике. Больше информации
В наших тестах мы используем ДНК легкой фракции клеток крови. Ее очень легко получить с помощью обычной настольной центрифуги и солевого раствора. Никаких дополнительных реагентов и минимальное время обработки. Дополнительная информация
Новый тип ферментов — Метилзависимые ДНК эндонуклеазы
Метилзависимые сайт-специфичные ДНК эндонуклеазы (МД эндонуклеазы) — новый класс ферментов открытый в SibEnzyme (Россия).
Эти ферменты очень напоминают обычные рестриктазы по своим биохимическим свойствам. Принципиальное же их отличие — они режут только метилированную ДНК. Это свойство делает МД-эндонуклеазы очень удобным инструментом для эпигенетического анализа.
В настоящее время коммерчески доступно 9 различных МД-эндонуклеаз. Сайты узнавания этих ферментов отличаются не только по структуре, но и по паттерну метилирования. Ко всему прочему они могут разрезать ДНК в сайте узнавания различными способами.
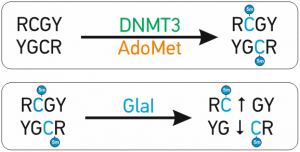
Особым свойством обладает фермент GlaI — его сайт узнавания R(5mC)GY является продуктом de novo метилирования метилазы DNMT3. Это свойство превращает GlaI в удобный инструмент для эпигенетической ДНК диагностики.
Известные применения:
- GLAD-ПЦР анализ — детекция наличия сайта R(5mC)GY в заданной точке
- GlaI-ПЦР анализ — количественное определение уровня метилирования в заданной точке
- EpiSeq — картирование положений и частоты метилирования сайтов RCGY во всем геноме
Заказывайте МД эндонуклеазы на сайте SibEnzyme или напишите нашим менеджерам на адрес nsk@sibenzyme.ru для получения дополнительной информации.
Определение метилирования ДНК в заданной точке без бисульфитной обработки: GLAD-ПЦР анализ
Два новых метода для анализа метилирования ДНК без бисульфитной обработки основаны на МД-эндонуклеазе GlaI. Методы позволяют определять наличие даже нескольких молекул метилированной ДНК (сайта R(5mC)GY) или неметилированной ДНК (сайта RCGY) в образце с избыточном количеством неметилированной или метилированной ДНК.
GLAD-ПЦР анализ (GlaI hydrolysis and Ligation Adapter Dependent PCR) позволяет определить наличие сайта 5′-R(5mC)GY-3′ в заданной точке ДНК человека или млекопитающего.
GLAD-ПЦР анализ включает три простых шага и выполняется в 1 пробирке [3] (см. анимацию ниже):
- Гидролиз ДНК ферментом GlaI
- подшивка универсального адаптера
- ПЦР в реальном времени с флюоресцентным зондом.
НЕ ТРЕБУЕТ БИСУЛЬФИТНОЙ ОБРАБОТКИ ДНК!
Новый метод может использоваться для определения метилирования целевого сайта в минимальных количествах (5 и более копий) в условиях присутствия фонового количества неметилированных сайтов.
Метод может применяться для ДНК, полученной из различных источников: тканей, биопсий, крови, плазмы, сыворотки и т.д.
GLAD-ПЦР анализ
Аберрантное de novo метилирование ДНК осуществляется ДНК-метилтрансферазами DNMT3A и DNMT3B. Эти ферменты распознают сайт 5’-RCGY-3’, а их продуктом является сайт 5’-R(5mC)GY-3’/3’YG(5mC)R-5’[1]. Открытая ранее сайт-специфическая метил-зависимая ДНК эндонуклеаза (MD-эндонуклеазы) GlaI узнает именно эту последовательность 5’-R(5mC)↓GY-3’/3’YG↓(5mC)R-5’ и разрезает ее в местах, указанных стрелками [2]












































Инновации и преимущества
В сравнении с традиционными методами анализа метилирования ДНК GLAD-ПЦР анализ:
- Простой – 3 простых шага в одной пробирке, требует обычную РВ-ПЦР машину
- Быстрый — всего 3-4 часа
- Чувствительный – определяет от 5 копий метилированного сайта в образце
- Надежный — ложно-положительные результаты отсутствуют
- Количественные — существуют протоколы для качественного и количественного анализа
Все эти факторы делают GLAD-ПЦР анализ очень удобным для эпигенетической ДНК диагностики
Ограничения традиционных методов
Главными проблемами при метилчувствительной ПЦР (MSP), основанной на бисульфитной конверсии ДНК, являются неполная конверсия, что ведет к ложно положительным результатам, или деградация ДНК при избыточной обработке.
Методы анализа, основанные на метил-чувствительных эндонуклеазах рестрикции таких как пара MspI-HpaII дают положительный результат только при отсутствии гидролиза HpaII. Таким образом, этот метод является косвенным. Кроме того, анализируемый сайт является второстепенным и не связан с de novo метилированием ДНК, происходящем при возникновении различных заболеваний.
Праймеры и флюоресцентные зонды
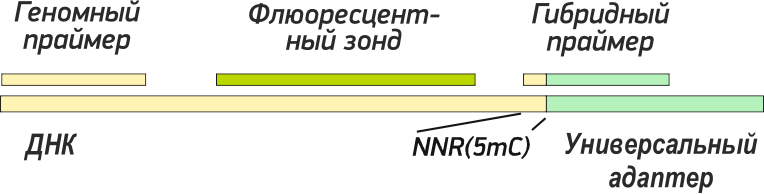
Для проведения GLAD-ПЦР анализа используется флюоресцентный зонд и 2 праймера: геномный и гибридный.
Геномный праймер и зонд подбираются как обычно в окрестности исследуемого сайта 5’-R(5mC)↓GY-3’ [4].
Гиридный праймер представляет собой последовательность 5’-CCTGCTCTTTCATCGGYNN-3’, где 5’ конец пятнадцатинуклеотидного праймера соответствует универсальному адаптеру, а четырехнуклеотидная часть на 3’ конце (подчеркнуто) соответствует последовательности ДНК в точке гидролиза GlaI. Подобная структура подразумевает существование 32 вариантов гибридного праймера соответсвующих различным последовательностям NNR(5mC)↓GY в точке гидролиза GlaI .
Все праймеры и зонды могут быть заказаны в компании SibEnzyme вместе с набором для GLAD-ПЦР анализа.
В качестве примера использования метода GLAD-ПЦР в наборе приведены протоколы для двух R(5mC)GY сайтов в человеческом геноме.
Наборы праймеров и зондов для этих двух R(5mC)GY сайтов включают:
- «Контрольная смесь для URB1 (праймеры + флюоресцентный зонд)» предназначена для анализа сайта A(5mC)GT в регуляторной области гена URB1. Сайт расположен (в соответствии с геномной сборкой GRCh38/hg38) в позиции 32334291-32334294 в 21 хромосоме.
- «Контрольная смесь для CEBPD (праймеры + флюоресцентный зонд)» предназначена для анализа сайта G(5mC)GC в регуляторной области гена CEBPD. Сайт расположен (в соответствии с геномной сборкой GRCh38/hg38) в позиции 47738502-47738505 в 8 хромосоме.
Набор для GLAD-ПЦР анализа

Для удобства разработчиков в компании SibEnzyme был подготовлен набор для проведения 200 или 1000 реакций GLAD-ПЦР в 2/10 96-луночных ПЦР планшетах. Набор включает все необходимые реагенты кроме праймеров и зондов. Последовательность праймеров и зонда зависит от структыры ДНК в окрестности исследуемого сайта RCGY .
Для реакции необходимо:
- исследуемый образец ДНК
- геномный праймер и зонд, подобранные под анализируемый RCGY сайт
- гибридный праймер — соответствующий структуре ДНК в точке гидролиза GlaI.
Набор для GLAD-ПЦР анализа можно заказать на сайте компании SibEnzyme или связаться с менеджерами компании по адресу order@sibenzyme.ru.
Подробная информация о наборе приведена в Инструкции по применению набора для GLAD-ПЦР анализа
Определение метилирования ДНК без бисульфитной обработки: GlaI-ПЦР анализ
Два новых метода для анализа метилирования ДНК без бисульфитной обработки основаны на МД-эндонуклеазе GlaI. Методы позволяют определять наличие даже нескольких молекул метилированной ДНК (сайта R(5mC)GY) или неметилированной ДНК (сайта RCGY) в образце с избыточном количеством неметилированной или метилированной ДНК.
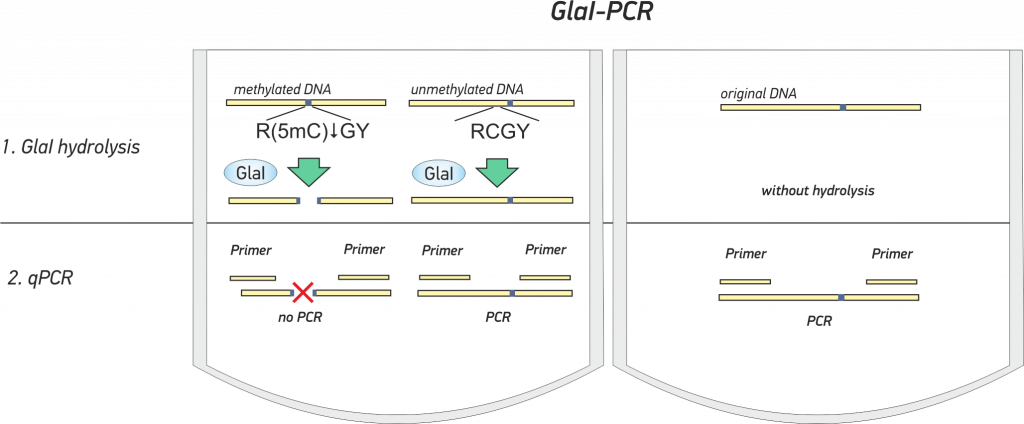
GlaI-анализ позволяет определять наличие сайта 5′-RCGY-3′ в заданной точке ДНК человека или млекопитающих.
GlaI-ПЦР анализ включает два шага и осуществляется в одной пробирке[3] (см. анимацию ниже):
- Гидролиз исследуемой ДНК с помощью фермента GlaI
- Парные реакции ПЦР в реальном времени с оригинальной и гидролизованной ДНК и сравнение из результатов
НЕ ТРЕБУЕТ БИСУЛЬФИТНОЙ ОБРАБОТКИ ДНК!
Метод GlaI-ПЦР анализа используется для определения минимальных количеств неметилированных сайтов 5′-RCGY-3′ в образце ДНЦ человека или млекопитающих в присутствии избыточного количества метилированных сайтов. Метод является удобным инструментом для эпигенетической ДНК диагностики и может использоваться в клинической практике.
Метод можно использовать для анализа препаратов ДНК из различных источников: тканей, биопсий, крови, плазмы, сыворотки и т.д.
GlaI-ПЦР анализ
Аберрантное de novo метилирование ДНК осуществляется ДНК-метилтрансферазами DNMT3A и DNMT3B. Эти ферменты распознают сайт 5’-RCGY-3’, а их продуктом является сайт 5’-R(5mC)GY-3’/3’YG(5mC)R-5’[1]. Открытая ранее сайт-специфическая метил-зависимая ДНК эндонуклеаза (MD-эндонуклеазы) GlaI узнает именно эту последовательность 5’-R(5mC)↓GY-3’/3’YG↓(5mC)R-5’ и разрезает ее в местах, указанных стрелками [2]





































Преимущества метода
В сравнении с другими методами анализа метилирования ДНК у метода GlaI-ПЦР анализа есть много преимуществ:
- Простота – 2 простых шага, требуется только оборудования для ПЦР в реальном времени
- Скорость – всего 2-3 часа для проведения анализа в 96 луночном планшете
- Чувствительность – позволяет обнаружить от 5 молекул неметилированной ДНК
- Количественный метод — может быть использован для определения процента метилирования сайта
Все эти факторы делают GlaI-ПЦР анализ очень привлекательным для эпигенетической ДНК диагностики.
Ограничения традиционных методов
Главными проблемами при метилчувствительной ПЦР (MSP), основанной на бисульфитной конверсии ДНК, являются неполная конверсия, что ведет к ложно положительным результатам, или деградация ДНК при избыточной обработке.
Метод GlaI-ПЦР анализа лишен этих недостатков
Праймеры и флюоресцентные зонды
Для GlaI-ПЦР анализа, как и для любой другой реакции ПЦР требуется флюоресцентно-меченый зонд и 2 праймера.
Праймеры и зонд подбираются как обычно в окрестностях исследуемого сайта 5’-RCGY-3’ [4].
Все праймеры и зонды могут быть заказаны в компании SibEnzyme вместе с набором для GlaI-ПЦР анализа.
В качестве примера использования метода GlaI-ПЦР в наборе приведен протокол для анализа сайта ACGС в регуляторной области гена RARb в человеческом геноме.
Наборы праймеров и зондов для анализа этого сайта, а также образцы ДНК для проведения контрольного эксперимента включены в набор.
Набор для GlaI-ПЦР анализа
Для удобства разработчиков в компании SibEnzyme был подготовлен набор для проведения 200 или 1000 реакций GlaI-ПЦР в 2/10 96-луночных ПЦР планшетах. Набор включает все необходимые реагенты кроме праймеров и зондов. Последовательность праймеров и зонда зависит от структыры ДНК в окрестности исследуемого сайта RCGY .
Для реакции необходимо:
- исследуемый образец ДНК
- геномные праймеры и зонд, подобранные под анализируемый RCGY сайт
Набор для GlaI-ПЦР анализа можно заказать на сайте компании SibEnzyme или связаться с менеджерами компании по адресу order@sibenzyme.ru.
Подробная информация о наборе приведена в Инструкции по применению набора для GlaI-ПЦР анализа
EpiSeq — новый метод пробоподготовки к NGS для построения профилей de novo метилирования ДНК
При разработке новых эпигенетических диагностикумов главный интерес для исследователей представляют сайты de novo метилирования ДНК в геноме — потенциальные маркеры метилирования. Известно, что de novo метилирование ДНК осуществляется метилтрансферазами DNMT3A и DNMT3B. Эти ферменты распознают и метилируют сайт 5’-RCGY-3’ с формировнием сайта 5’-R(5mC)GY-3’/3’YG(5mC)R-5’[1]. Т.е. существует всего 4 варианта сайта R(5mC)GY: A(5mC)GC, A(5mC)GT, G(5mC)GC, G(5mC)GT.
Новый метод пробоподготовки к NGS секвинированию — EpiSeq, разработанный в компании SibEnzyme позволяет картировать положение и частоту сайтов R(5mC)GY во всем геноме за 4 шага:
- Фрагментация ДНК: гидролиз ДНК ферментом GlaI с образованием тупых концов
- Отбор фрагментов ДНК: 150-700 п.н.
- Парноконцевое 75-150 п.н. NGS секвенирование на Illumina с использованием обычных реагентов
- Мэппинг прочитанных фрагментов на референсный геном. Концы фрагментов соответствуют позициям сайтов R(5mC)GY
Детальное описание метода приведено в публикациях [12, 14]
Преимущества метода
- Простота — в привычном протоколе требуется только заменить ультразвуковую фрагментацию на гидролиз GlaI, концы фрагментов при этом будут соответствовать сайтам метилирования
- для построения одного профиля достаточно уже 4-6 Gb данных; 15-20 профилей за 1 запуск HiSeq
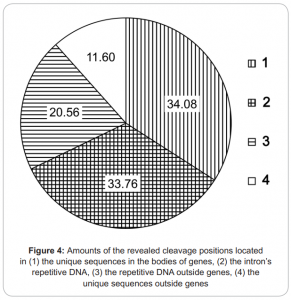
- покрытие метода составляет около 3.5 млн из 7 млн сайтов de novo метилирования ДНК в геноме
Ограничения традиционных методов
На сегодня наиболее полные данные могут быть получены при использовании метода полногеномного бисульфитного секвенирования (WGBS). Однако, данный метод является довольно дорогим и технологически сложным, возможно поэтому он не нашел широкого применения.
На практике используется более простой и дешевый метод RRBS (reduced representation bisulfite sequencing), который подразумевает секвенирование коротких, обработанных бисульфитом фрагментов MspI, тем не менее покрывающих достаточно большое количество CpG островков.
Также свою нишу заняли ДНК чипы для анализа конвертированной ДНК. Например, набор Infinium MethylationEPIC BeadChip Kit позволяющий определять статус метилирования около 850,000 сайтов CG в одном эксперименте. Однако, покрытие сайтов на таком чипе детерминировано и субъективно, к тому же метод достаточно дорог.
Общим недостатком методов, базирующихся на бисульфитной обработке ДНК является неполная конверсия ДНК или наоборот ее деградация в ходе химической обработки. Кроме того, они требуют проведения дополнительных этапов пробоподготовки, трудоемки и трудновоспроизводимы.
В настоящее время набор EpiSeq готовится к продаже, фермент GlaI коммерчески доступен. Заказать GlaI можно на сейте SibEnzyme или связавшись с менеджерами SibEnzyme по электронной почте order@sibenzyme.ru.
Выделение ДНК из легкой фракции клеток крови
В своих исследованиях мы используем ДНК из легкой фракции клеток крови. Получить эту фракции очень просто — потребуется только настольная центрифуга, солевой раствор и минимальное количество манипуляций с кровью. Эта простота и надежность метода позволяет избежать большинства проблем и ошибок медицинского персонала, возникающих при работе с внеклеточной или опухолевой ДНК крови. См. подробности использования метода [13].
Участие в конференциях, патенты и публикации
Научные конференции:
- Cancer Epigenetics 2017, Осака, Япония, октябрь 2017, постерная презентация Study of DNA methylation associated with Lung Cancer: GLAD-PCR Assay of R(5mC)GY sites
- Cancer Epigenetics 2017, Осака, Япония, октябрь 2017, постерная презентация Comparative analysis of RCGY sites methylation in the genomes of Raji and U-937 malignant and normal lung fibroblast cell lines
- Global Cancer Summit 2017, Куала Лумпур, Малайзия, март 2017, выступление GLAD-PCR assay of R(5mC)GY sites in aberrantly methylated regulation regions of tumor-suppression genes in lung cancer
- 43th ISOBM annual congress, Чикаго, США, сентябрь 2016, выступление GLAD-PCR assay of DNA methylation markers associated with colorectal cancer
- Global Cancer Summit 2015, Бангалор, Индия, выступление “GLAD-PCR assay of R(5mC)GY sites in aberrantly methylated regulation regions of tumor-suppression genes in colorectal cancer“
- Cancer Epigenomics 2015, Сан-Франциско, США, постерная презентация “Determination of R(5mC)GY sites in regulation regions of tumor-suppression genes by GLAD-PCR assay in colorectal cancer“
- Cell Symposia Cancer Epigenomics, Барселона, Испания, октябрь 2013, постерная презентация GLAD PCR analysis of aberrant DNA methylation in cancer
Публикации и патенты:
- Handa, V., and Jeltsch A. “Profound sequence preference of Dnmt3a and Dnmt3b mammalian DNA methyltransferases shape the human epigenome” // 2005, J. Mol. Biol. 348, 1103-1112
- Tarasova G.V., Nayakshina T.N., Degtyarev S.Kh. Substrate specificity of new methyl-directed DNA endonuclease GlaI // BMC Molecular Biology 2008, 9:7
- Kuznetsov V.V., Abdurashitov M.A., Akishev A.G., Degtyarev S.Kh. Method of determining nucleotide sequence Pu(5mC)GPy at predetermined position of continuous DNA // Patent RU 2525710 C1
- A.G. Akishev, M.A. Abdurashitov, V.L. Sitko, N.A. Netesova, I.F. Radaeva, E.A. Nechaeva, S.Kh. Degtyarev “GLAD-PCR assay of selected R(5mC)GY sites in URB1 and CEPBD genes in human genome” // Res J Pharm Biol Chem Sci, 8(1): pp.465-475, 2017
- A.A. Evdokimov , N.A. Netesova , N.A. Smetannikova , M.A. Abdurashitov, A.G. Akishev , B.S. Malyshev , E.S. Davidovich , V.V. Fedotov , V.V. Kuznetsov , Y.D. Ermolaev , A.B. Karpov , A.E. Sazonov , R.M. Tahauov, S.Kh. Degtyarev GLAD-PCR Assay of DNA Methylation Markers Associated with Colorectal Cancer // Biol Med (Aligarh) , 8:7(2016)
- A.A. Evdokimov, N.A. Netesova, N.A. Smetannikova, M.A. Abdurashitov, A.G. Akishev, E.S. Davidovich, Yu.D. Ermolaev, A.B. Karpov, A.E. Sazonov, R.M. Tahauov, S.Kh. Degtyarev Application of GLAD-PCR assay for determination of the methylation sites in the regulatory regions of tumor-supressors gene ELMO1 and ESR1 in colorectal cancer // Problems in oncology, #1, 2016 p.116-120
- Alexander G. Akishev, Danila A. Gonchar, Murat A. Abdurashitov and Sergey Kh. Degtyarev Epigenetic typing of human cancer cell lines by BlsI- and GlaI-PCR assays // “Ovchinnikov bulletin of biotechnology and physical and chemical biology” V.7, No 3, pp 5-16, 2011
- D. A. Gonchar, A. G. Akishev, S. Kh. Degtyarev BlsI- and GlaI-PCR assays – a new method of DNA methylation study // “Ovchinnikov bulletin of biotechnology and physical and chemical biology” V.6, No 1, pp 5-12, 2010
- V.A. Chernukhin, T.N. Najakshina, M.A. Abdurashitov, J.E. Tomilova, N.V. Mezentzeva, V.S. Dedkov, N.A. Mikhnenkova, D.A. Gonchar, S.Kh. Degtyarev A novel restriction endonuclease GlaI recognizes methylated sequence 5’-G(5mC)^GC-3’ // Biotechnologia (russ.). 2006. N 4. P. 31-35
- A.A. Evdokimov, M.V. Kulak, N.A. Netesova, N.A. Smetannikova, M.A., Abdurashitov, A.G. Akishev, B.S. Malyshev, E.S. Davidovich, V.V. Fedotov, A.B. Karpov, S.Kh. Degtyarev GLAD-PCR Assay of DNA Methylation Markers Associated with Colorectal Cancer // Tumor Biology, Vol.37, supplement 1, Sept. 2016, s6-s7
- Abdurashitov M.A., Tomilov V.N., Gonchar D.A., Dubinin E.V., Degtyarev S.Kh. “Mapping positons of methylated sequences Pu(5mC)GPy along continuous DNA for epigenetic profile creation and identification of aberrant DNA methylation sites” // Patent RU 2586502 C1
- Abdurashitov M.A., Tomilov V.N., Gonchar D.A., Kuznetsov V.V., Degtyarev S.Kh. Mapping of R(5mC)GY Sites in the Genome of Human Malignant Cell Line Raji // Biol Med (Aligarh) Volume 7, Issue 4, BM-135-15 (2015)
- А.Г. Акишев, Н.А. Нетесова, М.А. Абдурашитов, И.В. Вихлянов, М.К. Никитин, А.Б. Карпов, С.Х. Дегтярев (2019) GlaI-ПЦР анализ метилирования сайта ACGC в хp11: 65647266 в препаратах ДНК клеток крови в норме и на ранних стадиях рака молочной железы, Эпигенетич ДНК диагност, том 2019(1), DOI: 10.26213/SE.2019.66.12.001
- Abdurashitov M.A., Tomilov V.N., Gonchar D.A., Snezhkina A.V., Krasnov G.S., Kudryavtseva A.V., Degtyarev S.Kh. Comparative Analysis Of RCGY Sites Methylation In Three Human Cell Lines. Epigenetic DNA diagnostics, vol.1, 2019 doi:26213/SE.2019.76.40116 Accessed 11 Nov 2019
Дополнительные PDF материалы для загрузки
- Methyl-directed DNA Endonucleases
- GLAD-PCR assay
- GLAD PCR assay Kit
- Early Cancer Detection
- Determination of R(5mC)GY sites in regulation regions of tumor suppression genes by GLAD-PCR assay in colorectal cancer